はじめてのMCMC (スライス・サンプリング)
こんにちはtatsyです。
MCMCの記事も終盤ですが、今回はスライス・サンプリングです。なお、これまでの記事はこちらです。
スライス・サンプリングとは?
スライス・サンプリングは前回紹介したハミルトニアン・モンテカルロと同様に拡張変数を用いるハイブリッド・モンテカルロの一種です。
スライス・サンプリングはこれまでのMCMCが抱えていた局所的に確率密度が高い部分を多くサンプルしてしまい、複数の不連続な台を持つような確率密度関数をうまくサンプルできない、という問題を解決しようとしています。
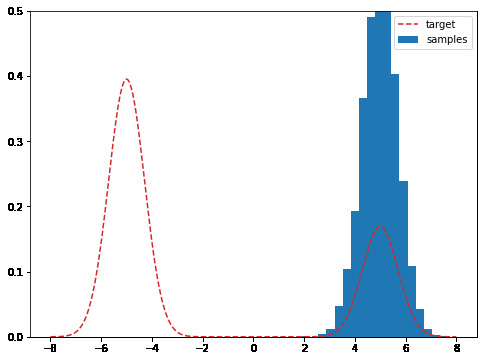
例えば、以下の画像に示すような二つのガウス分布が離れて存在しているような分布をM-H法でサンプリングしてしまうと、片方の山にサンプルが偏ってしまい、適切なサンプリングが行えていないことが分かります。
この問題は、今まで紹介したMCMCが主に今のサンプルの周辺から提案サンプルを得ていたことに起因します。この問題を解決するため、スライス・サンプリングは近傍に限らず、山が高いところから新しいサンプルを得ることを目指します。
この目的のために、新たに基準の山の高さを表す変数$u$を導入します。この$u$は現在のサンプル$x$における目的の確率密度関数の値より低い値をランダムに抽出します。
あとは、確率密度全体から$u$より高い$p(x)$を持つ$x$から一様に新たなサンプルを得ます。しかしながら、このある高さ以上の場所から一様にサンプルを抽出するというのは一般に容易では有りません。なぜなら、一般にはある高さ以上の確率密度を持つ場所というのはサンプルが容易な関数の形はしていないし、そもそもが範囲を正確に特定できなかったり、非連結であったりする場合が多いからです。
そこで、スライス・サンプリングでは現在の位置$x^t$を中心として、その両側に広がる領域$[x_{min}, x_{max}]$を考え、その領域内で棄却サンプリングを行うことで、上記の$u$より高い部分からの擬似的な一様サンプリングを実現します。最初に$x$を中心として正負それぞれの方向に適当な幅だけ離れた$x_{min}, x_{max}$を取り、その両端における確率密度が$u$を下回るまで$x_{min}, x_{max}$のそれぞれを広げていきます。
広げ終わったら、$[x_{min}, x_{max}]$の中から一様サンプリングによって新たな提案サンプル$x^{t+1}$を得ます。もちろん、適当な幅で$x_{min}, x_{max}$を広げていった場合、たまたまその区間内に$u$より低い高さを持つ$x$が存在するかもしれないので、もしそのような$x$が見つかった場合には、$x_{min}$と$x_{max}$のいずれかを$x$として、区間が狭くなるように更新します。
上記の操作を$u$より高い高さを持つ$x^{t+1}$が見つかるまで繰り返すことで、サンプルの更新を行う、というのがスライスサンプリングです。
以下の例に示すとおり、十分に$x_{min}$ないし$x_{max}$を広げる幅を大きくとってあげれば、離れた領域において高い確率密度を取るような目的分布に対しても、所望のサンプリングを行うことができています。
スライス・サンプリングが上手くいく理由
上記の結果から、スライス・サンプリングが正しく目的分布からのサンプリングを実現していることは分かります。ですが、なぜ上記の方法で、正しくサンプリングが行えるのかをもう少し詳しく見ていきます。
まずはじめに、とある$u$より$p(x)$が大きいときに1を取る指示関数$\varphi_{p(x) > u} (x)$を考えます。すると、$x^t$から別の$x^{t+1}$へとサンプルが遷移する確率$q(x^{t+1}|x^t)$は確率密度関数の定義域全体を$\Omega$として、次のように表せます。 $$ q(x^{t+1} | x^t) = \int_0^{p(x^t)} \int_\Omega \varphi_{p(x^{t+1}) > u} dx^{t+1} du $$ 細かい話は考えないこととして、上記の積分が順序変更可能であるとすると、 $$ q(x^{t+1} | x^t) = \int_\Omega \int_0^{p(x^t)} \varphi_{p(x^{t+1}) > u} du dx^{t+1} $$ となります。この時、あらためて$du$が確率測度である、すなわち各$x^t$について $$ \int_0^{p(x^t)} du = 1 $$ を満たすことに注意すると、上記の二重積分の内側の積分は各$x^t$と$x^{t+1}$のペアについて、$p(x^{t}) < p(x^{t+1})$ならば1を、反対に$p(x^t) > p(x^{t+1})$ならば$\frac{p(x^{t+1})}{p(x^t)}$を取ることが分かります。
従って、式を整理すれば、 $$ q(x^{t+1} | x^t) = \int_\Omega \min \left[ 1, \frac{p(x^{t+1})}{p(x^t)} \right] dx^{t+1} $$ となります。同様に、 $$ q(x^t | x^{t+1}) = \int_\Omega \min \left[ 1, \frac{p(x^{t})}{p(x^{t+1})} \right] dx^{t} $$ となりますが、これを詳細釣り合いの式に代入してみると、 $$ \begin{aligned} p(x^t) q(x^{t+1} | x^t) &= \int_\Omega \min \left[ p(x^t), p(x^{t+1}) \right] dx^{t+1} \\ &= \int_\Omega \min \left[ p(x^t), p(x^{t+1}) \right] dx^{t} \\ &= p(x^{t+1}) \int_\Omega \min \left[ 1, \frac{p(x^t)}{p(x^{t+1})} \right] dx^{t} \\ &= p(x^{t+1}) q(x^t | x^{t+1}) \end{aligned} $$ となり、詳細釣り合いの条件が成り立ちます。以上の理由からスライス・サンプリングにより正しく目的の確率密度分布がサンプリングできることが分かります。
ただし、実際には$u < p(x^{t+1})$を満たす全ての$x^{t+1}$から一様サンプリングすることはできず、擬似的に$[x_{min}, x_{max}]$の間でサンプリングをしているため、おそらく、実用的には「近似的な」サンプリングしか行えないと思います (私自身はあまりわかっていませんが、もしご存知の方がいればご教示いただければ幸いです)。
このことから、なるべく$x_{min}, x_{max}$を広げる幅 (コード中におけるself.w)は大きめに設定するほうが望ましいのですが、大きくしすぎると、新しいサンプルが頻繁に$p(x^{t+1}) < u$となって棄却されてい舞うため、サンプリングの効率が落ちてしまいます。このあたりはスライス・サンプリングのトレードオフ、ということになります。
まとめ
スライス・サンプリングは多次元のものに適用しようとする場合には、各次元ごとに上記の処理を実行して新たなサンプル点を得ることになりますので、他の手法に比べるとやや実装は煩雑になります。
また、スライス・サンプリングの特徴として、M-H法などのように提案分布を用いないので、提案分布の性能にサンプリング結果が依存しづらいということが挙げられます。とはいうものの、実際には$x_{min}, x_{max}$の更新幅を適切に設定する必要は生じるため、どちらが一概に良いとは言い切れない部分もあります。
以上、簡単になりますが、がスライス・サンプリングの紹介です。全4回の「はじめてのMCMC」も今回で最後です。最後までお読みいただき、ありがとうございました。